Unter Genexpression verstehen wir die Transkription eines Gens in mRNA und seine anschließende Translation in Protein. Die Genexpression wird hauptsächlich auf der Ebene der Transkription kontrolliert, hauptsächlich als Folge der Bindung von Proteinen an bestimmte Stellen auf der DNA. 1965 erhielten Francois Jacob, Jacques Monod und Andre Lwoff den Nobelpreis für Medizin für ihre Arbeit, die die Idee unterstützt, dass die Kontrolle der Enzymspiegel in Zellen durch Transkription von DNA reguliert wird., tritt durch Regulierung der Transkription auf, die entweder induziert oder unterdrückt werden kann. Diese Forscher schlugen vor, dass die Produktion des Enzyms durch ein „Operon“ gesteuert wird, das aus einer Reihe verwandter Gene auf dem Chromosom besteht, die aus einem Operator, einem Promotor, einem Regulator-Gen und Strukturgenen bestehen.
- Die Strukturgene enthalten den Code für die zu produzierenden Proteinprodukte. Die Regulierung der Proteinproduktion wird weitgehend durch die Modulierung des Zugangs der RNA-Polymerase zu dem transkribierten Strukturgen erreicht.,
- Das Promotorgen kodiert nichts; es ist einfach eine DNA-Sequenz, die anfängliche Bindungsstelle für RNA-Polymerase ist.
- Das Operatorgen ist ebenfalls nicht kodierend; Es ist nur eine DNA-Sequenz, die die Bindungsstelle für den Repressor ist.
- Das Regulator-Gen kodiert für die Synthese eines Repressormoleküls, das an den Operator bindet und die RNA-Polymerase daran hindert, die Strukturgene zu transkribieren.
Das Operatorgen ist die Sequenz nicht transkribierbarer DNA, die die Repressor-Bindungsstelle ist., Es gibt auch ein Regulatorgen, das für die Synthese eines Repressormoleküls kodiert, das an den Operator
- Beispiel für induzierbare Transkription bindet: Das Bakterium E. coli hat drei Gene, die für Enzyme kodieren, die es ermöglichen, Laktose (einen Zucker in Milch) zu spalten und zu metabolisieren. Der Promotor ist die Stelle auf DNA, an der RNA-Polymerase bindet, um die Transkription zu initiieren. Die Enzyme sind jedoch üblicherweise in sehr geringen Konzentrationen vorhanden, da ihre Transkription durch ein Repressorprotein gehemmt wird, das von einem Regulator-Gen produziert wird (siehe den oberen Teil der Abbildung unten)., Das Repressorprotein bindet an die Operatorstelle und hemmt die Transkription. Wenn jedoch Laktose in der Umgebung vorhanden ist, kann sie an das Repressorprotein binden und es inaktivieren, wodurch die Blockade wirksam beseitigt und die Transkription der für die Synthese dieser Gene erforderlichen Boten-RNA ermöglicht wird (unterer Teil der Abbildung unten).

- Beispiel Repressible Transkription: E. coli benötigen die Aminosäure tryptophan, und die DNA in E. coli hat auch Gene für die Synthese von es., Diese Gene transkribieren im Allgemeinen kontinuierlich, da das Bakterium Tryptophan benötigt. Wenn die Tryptophankonzentrationen jedoch hoch sind, wird die Transkription unterdrückt (ausgeschaltet), indem sie an ein Repressorprotein gebunden und aktiviert wird, wie unten dargestellt.
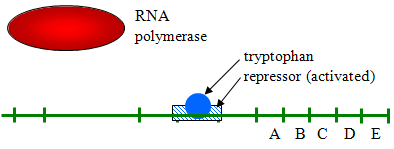
Kontrolle der Genexpression in Eukaryoten
Eukaryotische Zellen haben ähnliche Mechanismen zur Kontrolle der Genexpression, sind jedoch komplexer., Betrachten wir zum Beispiel, dass prokaryotische Zellen einer bestimmten Spezies alle gleich sind, aber die meisten Eukaryoten sind vielzellige Organismen mit vielen Zelltypen, so dass die Kontrolle der Genexpression viel komplizierter ist. Es überrascht nicht, dass die Genexpression in eukaryotischen Zellen durch eine Reihe komplexer Prozesse gesteuert wird, die in der folgenden Liste zusammengefasst sind.
- Nach der Befruchtung werden die Zellen im sich entwickelnden Embryo zunehmend spezialisiert, hauptsächlich durch Einschalten einiger Gene und Ausschalten vieler anderer., Einige Zellen in der Bauchspeicheldrüse sind beispielsweise auf die Synthese und Sekretion von Verdauungsenzymen spezialisiert, während andere Pankreaszellen (β-Zellen in den Langerhans-Inseln) auf die Synthese und Sekretion von Insulin spezialisiert sind. Jeder Zelltyp hat ein bestimmtes Muster exprimierter Gene. Diese Differenzierung
 in spezialisierte Zellen erfolgt weitgehend als Folge der Abschaltung der Expression der meisten Gene in der Zelle; Reife Zellen dürfen nur 3-5% der im Zellkern vorhandenen Gene verwenden.,
in spezialisierte Zellen erfolgt weitgehend als Folge der Abschaltung der Expression der meisten Gene in der Zelle; Reife Zellen dürfen nur 3-5% der im Zellkern vorhandenen Gene verwenden., - Die Genexpression in Eukaryoten kann auch durch Veränderungen in der DNA-Verpackung reguliert werden, wodurch der Zugang der Transkriptionsenzyme der Zelle (z. B. RNA-Polymerase) zur DNA moduliert wird. Die folgende Abbildung zeigt, dass Chromosomen eine komplexe Struktur haben. Die DNA-Helix ist um spezielle Proteine, sogenannte Histone, gewickelt, die in enge helikale Fasern gewickelt sind. Diese Fasern werden dann geschlungen und zu immer kompakteren Strukturen gefaltet, die, wenn sie vollständig gewickelt und kondensiert sind, den Chromosomen ihr charakteristisches Aussehen in der Metaphase
 verleihen.,
verleihen.,
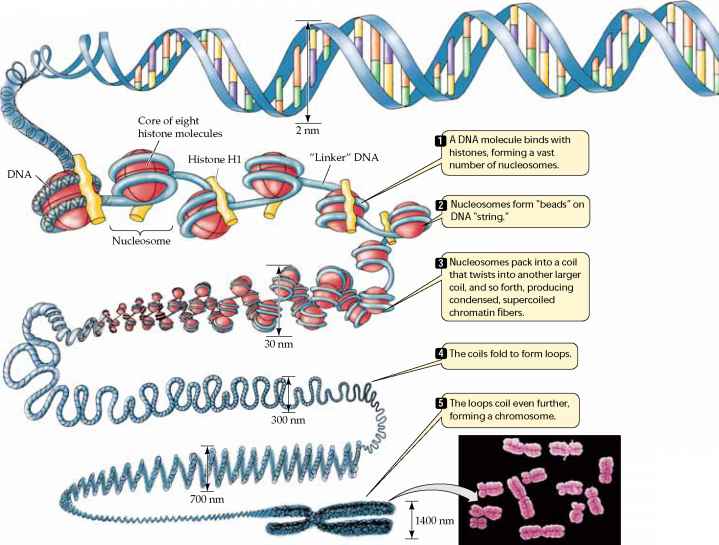
Quelle: http://www.78stepshealth.us/plasma-membrane/eukaryotic-chromosomes.html
- Ähnlich wie die oben beschriebenen Operonen für Prokaryoten verwenden Eukaryoten auch regulatorische Proteine, um die Transkription zu kontrollieren, aber jedes eukaryotische Gen hat sein eigener Satz von Kontrollen. Darüber hinaus gibt es viel mehr regulatorische Proteine in Eukaryoten und die Wechselwirkungen sind viel komplexer.,
- In Eukaryoten findet die Transkription innerhalb des membrangebundenen Kerns statt, und das anfängliche Transkript wird modifiziert, bevor es vom Kern zum Zytoplasma zur Translation auf dem Ribosom s transportiert wird. Das anfängliche Transkript in Eukaryoten hat kodierende Segmente (exons
 ) abwechselnd mit nicht kodierenden Segmenten (Introns)., Bevor die mRNA den Kern verlässt, werden die Introns durch einen Prozess namens RNA-Spleißen aus dem Transkript entfernt (siehe Grafik & Video unten), und zusätzliche Nukleotide werden zu den Enden des Transkripts hinzugefügt; Diese nicht kodierenden „Kappen“ und „Schwänze“ schützen die mRNA vor Angriffen durch zelluläre Enzyme und helfen bei der Erkennung durch die Ribosomen.,
) abwechselnd mit nicht kodierenden Segmenten (Introns)., Bevor die mRNA den Kern verlässt, werden die Introns durch einen Prozess namens RNA-Spleißen aus dem Transkript entfernt (siehe Grafik & Video unten), und zusätzliche Nukleotide werden zu den Enden des Transkripts hinzugefügt; Diese nicht kodierenden „Kappen“ und „Schwänze“ schützen die mRNA vor Angriffen durch zelluläre Enzyme und helfen bei der Erkennung durch die Ribosomen.,
Source: http://unmug.com/category/biology/organisation-control-of-genome/
![]()
- Die Variation der Langlebigkeit von mRNA bietet eine weitere Möglichkeit zur Kontrolle der Genexpression. Prokaryotische mRNA ist sehr kurzlebig, aber eukaryotische Transkripte können Stunden oder manchmal sogar Wochen dauern (z. B. mRNA für Hämoglobin in den roten Blutkörperchen von Vögeln).,
- Der Translationsprozess bietet zusätzliche Möglichkeiten zur Regulation durch viele Proteine. Zum Beispiel wird die Translation von Hämoglobin-mRNA gehemmt, es sei denn, eisenhaltiges Häm ist in der Zelle vorhanden.
- Es gibt auch Möglichkeiten für „posttranslationale“ Kontrollen der Genexpression in Eukaryoten. Einige übersetzte Polypeptide (Proteine) werden von Enzymen in kleinere, aktive Endprodukte geschnitten. wie in der folgenden Abbildung dargestellt, die die posttranslationale Verarbeitung des Hormons Insulin darstellt., Insulin wird zunächst als großer, inaktiver Vorläufer übersetzt; Eine Signalsequenz wird vom Kopf des Vorläufers entfernt, und ein großer zentraler Teil (die C-Kette) wird abgeschnitten, wobei zwei kleinere Peptidketten zurückbleiben, die dann durch Disulfidbrücken miteinander verbunden sind.Die kleinere endgültige Form ist die aktive Form von Insulin.
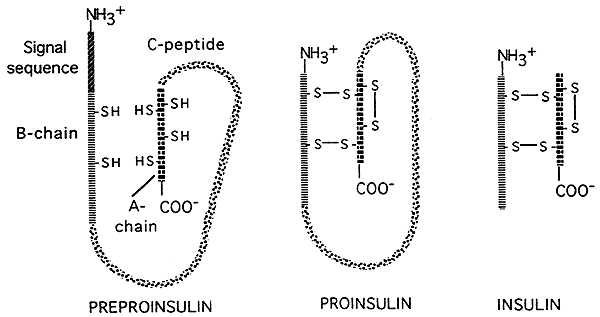
Quelle: http://www.nbs.csudh.edu/chemistry/faculty/nsturm/CHE450/19_InsulinGlucagon.htm
- Die Genexpression kann auch durch den Abbau der produzierten Proteine modifiziert werden., Zum Beispiel werden einige der am Zellstoffwechsel beteiligten Enzyme kurz nach ihrer Produktion abgebaut; Dies bietet einen Mechanismus, um schnell auf sich ändernde Stoffwechselanforderungen zu reagieren.
- Die Genexpression kann auch durch Signale anderer Zellen beeinflusst werden. Es gibt viele Beispiele, in denen ein Signalmolekül (z. B. ein Hormon) von einer Zelle an ein Rezeptorprotein auf einer Zielzelle bindet und eine Sequenz biochemischer Veränderungen (einen Signaltransduktionsweg) initiiert, die zu Veränderungen innerhalb der Zielzelle führen., Diese Änderungen können eine erhöhte oder verringerte Transkription umfassen, wie in der folgenden Abbildung dargestellt.
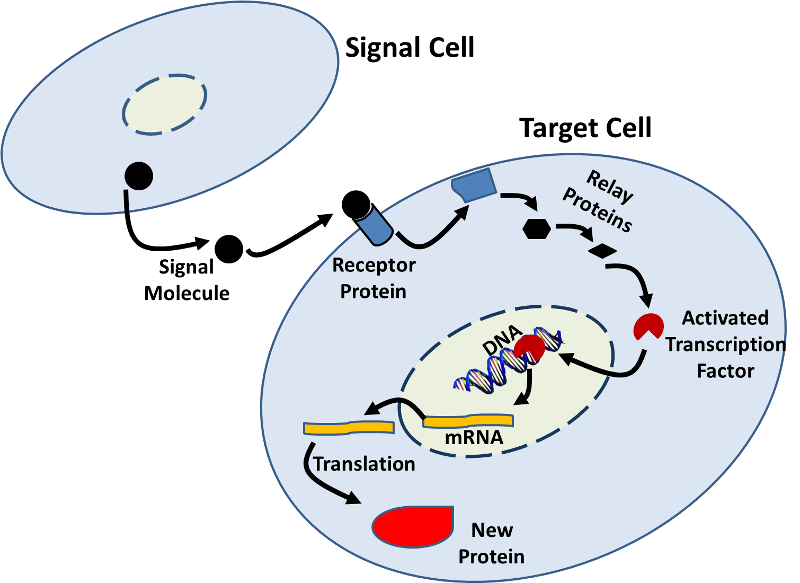
- Das RNA-Interferenzsystem (RNAi) ist ein weiterer Mechanismus, mit dem Zellen die Genexpression steuern, indem sie die mRNA-Translation abschalten. RNAi kann auch verwendet werden, um die Translation viraler Proteine abzuschalten, wenn eine Zelle mit einem Virus infiziert ist. Das RNAi-system hat auch das Potenzial, therapeutisch genutzt werden.,
RNAi
Einige RNA-Viren dringen in Zellen ein und führen doppelsträngige RNA ein, die die Zellmaschinerie verwendet, um neue Kopien von viraler RNA und viralen Proteinen herzustellen. Das RNA-Interferenzsystem (RNAi) der Zelle kann die Replikation der viralen RNA verhindern. Zuerst zerkleinert ein Enzym mit dem Spitznamen „Dicer“ jede doppelsträngige RNA, die es findet, in Stücke, die etwa 22 Nukleotide lang sind. Als nächstes binden Proteinkomplexe, die als RISC (RNA-Induced Silencing Complex) bezeichnet werden, an die Fragmente von doppelsträngiger RNA, wickeln sie auf und setzen dann einen der Stränge frei, während der andere erhalten bleibt., Der RISC-RNA-Komplex bindet dann an jede andere virale RNA mit Nukleotidsequenzen, die denen der an den Komplex angehängten RNA entsprechen. Diese Bindung blockiert die Translation viraler Proteine zumindest teilweise, wenn nicht sogar vollständig. Das RNAi-System könnte möglicherweise verwendet werden, um Behandlungen für defekte Gene zu entwickeln, die Krankheiten verursachen. Die Behandlung würde eine doppelsträngige RNA aus dem erkrankten Gen herstellen und in Zellen einführen, um die Expression dieses Gens zum Schweigen zu bringen., Eine illustrierte Erklärung von RNAi finden Sie im kurzen, interaktiven Flash-Modul unter http://www.pbs.org/wgbh/nova/body/rnai-explained.html
Das RNA-Interferenzsystem wird auch im folgenden Video von Nature Video ausführlicher erläutert.
![]()
![]()
zurück nach oben / vorherige Seite / nächste Seite