por expressão do gene queremos dizer a transcrição de um gene para o ARNm e sua subsequente tradução para a proteína. A expressão genética é controlada principalmente ao nível da transcrição, em grande parte como resultado da ligação de proteínas a locais específicos do ADN. Em 1965, François Jacob, Jacques Monod e Andre Lwoff compartilharam o Prêmio Nobel de medicina por seu trabalho apoiando a ideia de que o controle dos níveis de enzimas nas células é regulado pela transcrição do DNA., ocorre através da regulação da transcrição, que pode ser induzida ou reprimida. Estes pesquisadores propuseram que a produção da enzima é controlada por um “operão”, que consiste numa série de genes relacionados no cromossomo consistindo de um operador, um promotor, um gene regulador e genes estruturais.
- os genes estruturais contêm o código dos produtos proteicos a produzir. A regulação da produção de proteínas é largamente conseguida através da modulação do acesso da ARN polimerase ao gene estrutural que está a ser transcrito.,
- o gene promotor não codifica nada; é simplesmente uma sequência de ADN que é o local de ligação inicial para a ARN polimerase.
- o gene do operador também não é codificador; é apenas uma sequência de DNA que é o local de ligação para o compressor.
- o gene regulador codes for synthesis of a Compressor molecule that binds to the operator and blocks RNA polymerase from transcribing the structural genes.
o gene do operador é a sequência de ADN não transcribível que é o local de ligação do compressor., Existe também um gene regulador, que codifica para a síntese de uma molécula de compressor que se liga ao operador
- Exemplo de transcrição indutível: a bactéria E. coli tem três genes que codificam para enzimas que lhe permitem dividir e metabolizar a lactose (um açúcar no leite). O promotor é o local no ADN onde a ARN polimerase se liga para iniciar a transcrição. No entanto, as enzimas estão geralmente presentes em concentrações muito baixas, porque a sua transcrição é inibida por uma proteína repressora produzida por um gene regulador (ver a parte superior da figura abaixo)., A proteína do compressor liga-se ao local do operador e inibe a transcrição. No entanto, se a lactose está presente no ambiente, pode ligar-se à proteína do compressor e inactivá-la, removendo eficazmente o bloqueio e permitindo a transcrição do ARN mensageiro necessário para a síntese destes genes (parte inferior da figura abaixo).

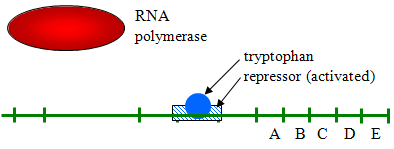
- Exemplo de Repressible Transcrição: E. coli necessidade de aminoácido, o triptofano, e o DNA em E. coli também tem genes para sintetizá-lo., Estes genes geralmente transcrevem continuamente uma vez que a bactéria precisa de triptofano. No entanto, se as concentrações de triptofano forem elevadas, a transcrição é reprimida (desligada) ligando-se a uma proteína repressora e activando-a como ilustrado abaixo.
Controlo da expressão genética em eucariotas
as células eucarióticas têm mecanismos semelhantes para controlo da expressão genética, mas são mais complexas., Considere, por exemplo, que as células procarióticas de uma determinada espécie são todas as mesmas, mas a maioria dos eucariontes são organismos multicelulares com muitos tipos de células, então o controle da expressão genética é muito mais complicado. Não surpreendentemente, a expressão genética nas células eucarióticas é controlada por uma série de processos complexos que são resumidos pela seguinte lista.após a fertilização, as células do embrião em desenvolvimento tornam-se cada vez mais especializadas, em grande parte ligando alguns genes e desligando muitos outros., Algumas células do pâncreas, por exemplo, são especializadas para sintetizar e secretar enzimas digestivas, enquanto outras células pancreáticas (células-β nas ilhotas de Langerhans) são especializadas para síntese e secretar insulina. Cada tipo de célula tem um padrão particular de genes expressos. Esta diferenciação em células especializadas ocorre em grande parte como resultado de desligar a expressão da maioria dos genes na célula; as células maduras só podem usar 3-5% dos genes presentes no núcleo da célula.,
em células especializadas ocorre em grande parte como resultado de desligar a expressão da maioria dos genes na célula; as células maduras só podem usar 3-5% dos genes presentes no núcleo da célula.,
 .,
.,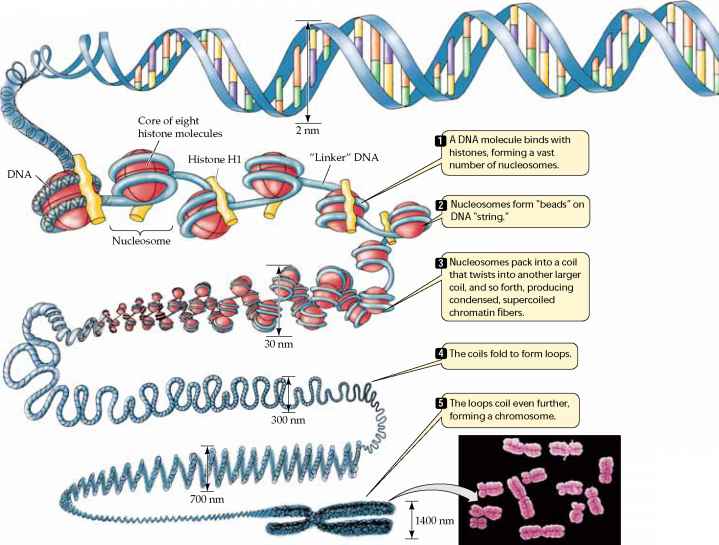
Fonte: http://www.78stepshealth.us/plasma-membrane/eukaryotic-chromosomes.html
- Semelhante para os operons descrito acima para procariotas, eucariontes, também, regulamentar o uso de proteínas para controlar a transcrição, mas cada eucarióticas gene tem seu próprio conjunto de controles. Além disso, existem muitas mais proteínas regulamentares nos eucariontes e as interacções são muito mais complexas.,
- Em eucariotas transcrição ocorre dentro da membrana dependente do núcleo, e o inicial de transcrição é modificado antes de ele é transportado do núcleo para o citoplasma para a tradução no ribossomo s. A inicial transcrição em eucariotas tem de codificação de segmentos (exões
 ), alternando com a não-codificadores de segmentos (íntrons)., Antes do mRNA deixa o núcleo, os íntrons são removidos a partir da transcrição por um processo chamado de RNA splicing (ver gráfico & vídeo abaixo), e extra nucleotídeos são adicionados para as extremidades da transcrição; estes não-codificadores de “caps” e “caudas” proteger o mRNA do ataque de celulares, enzimas e ajuda no reconhecimento, pelos ribossomos.,
), alternando com a não-codificadores de segmentos (íntrons)., Antes do mRNA deixa o núcleo, os íntrons são removidos a partir da transcrição por um processo chamado de RNA splicing (ver gráfico & vídeo abaixo), e extra nucleotídeos são adicionados para as extremidades da transcrição; estes não-codificadores de “caps” e “caudas” proteger o mRNA do ataque de celulares, enzimas e ajuda no reconhecimento, pelos ribossomos.,
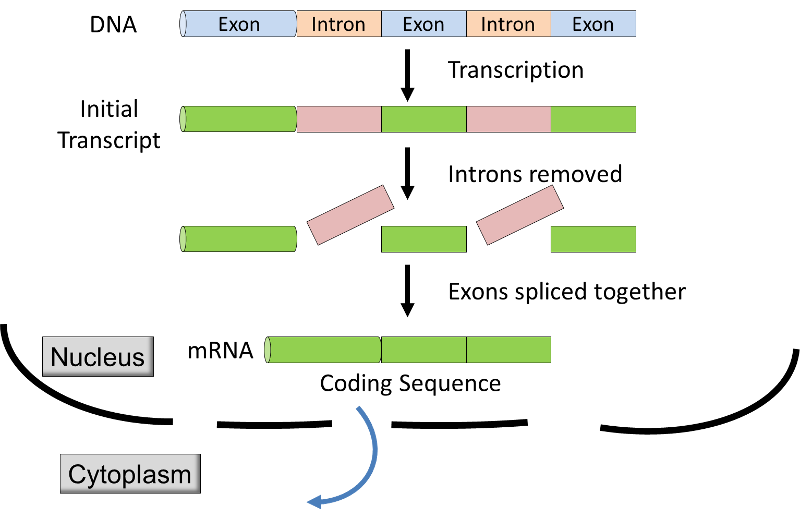
Fonte: http://unmug.com/category/biology/organisation-control-of-genome/
![]()
- Variação na longevidade de mRNA é mais uma oportunidade para o controle da expressão gênica. O ARNm procariótico é de curta duração, mas transcrições eucarióticas podem durar horas, ou até mesmo semanas (por exemplo, ARNm para hemoglobina nos glóbulos vermelhos das aves).,o processo de Tradução oferece oportunidades adicionais de regulação por muitas proteínas. Por exemplo, a tradução da hemoglobina mRNA é inibida a menos que o heme contendo ferro esteja presente na célula.existem também oportunidades para controlos” pós-translacionais ” da expressão genética em eucariotas. Alguns polipeptídeos traduzidos (proteínas) são cortados por enzimas em produtos finais mais pequenos e ativos. como ilustrado na figura abaixo, que retrata o processamento pós-Translacional da hormona insulina., A insulina é inicialmente traduzida como um grande precursor inativo; uma sequência de sinal é removida da cabeça do precursor, e uma grande porção central (a cadeia-C) é cortada, deixando duas cadeias peptídicas menores que são então ligadas umas às outras por pontes dissulfeto.A forma final mais pequena é a forma activa da insulina.
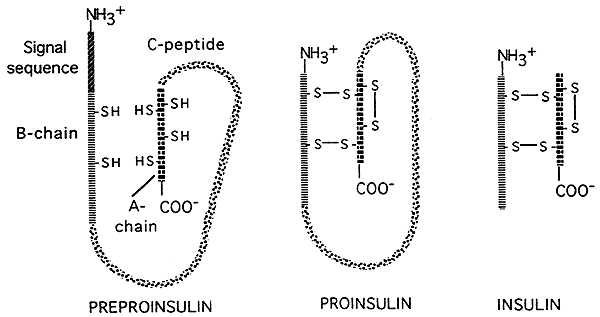
Fonte: http://www.nbs.csudh.edu/chemistry/faculty/nsturm/CHE450/19_InsulinGlucagon.htm
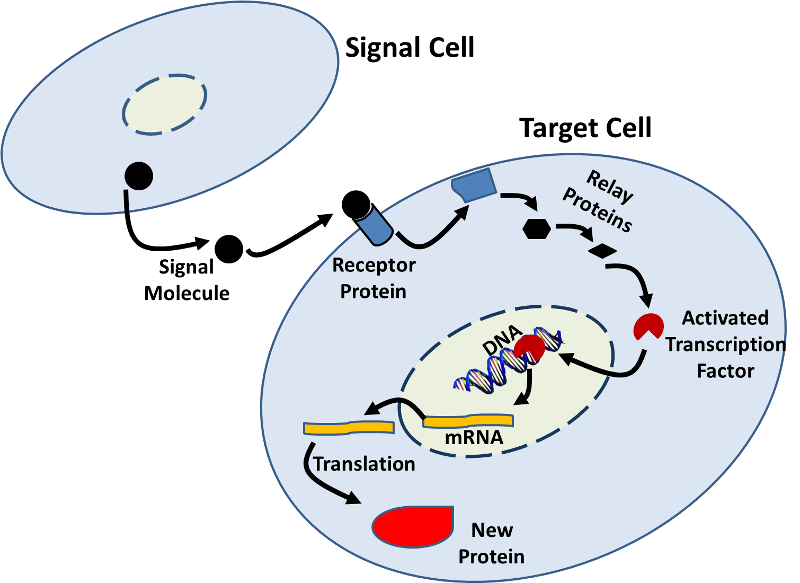
- a expressão do Gene também pode ser modificada pela decomposição de proteínas que são produzidas., Por exemplo, algumas das enzimas envolvidas no metabolismo celular são decompostas logo após serem produzidas, o que fornece um mecanismo para responder rapidamente às mudanças de demanda metabólica.a expressão do Gene pode também ser influenciada por sinais de outras células. Existem muitos exemplos em que uma molécula de sinal (por exemplo, uma hormona) de uma célula se liga a uma proteína receptora numa célula-alvo e inicia uma sequência de alterações bioquímicas (uma via de transdução de sinal) que resultam em alterações dentro da célula-alvo., Estas alterações podem incluir transcrição aumentada ou diminuída, como ilustrado na figura abaixo.
- RNA de Interferência do sistema (RNAi) é outro mecanismo pelo qual as células de controle de expressão gênica por desligar a tradução do mRNA. RNAi também pode ser usado para desligar a tradução de proteínas virais quando uma célula é infectada por um vírus. O sistema RNAi também tem o potencial de ser explorado terapeuticamente.,
RNAi
algum vírus de RNA invadirá as células e introduzirá RNA de cadeia dupla que usará a maquinaria das células para fazer novas cópias de RNA viral e proteínas virais. O sistema de interferência de RNA da célula (RNAi) pode prevenir a replicação do RNA viral. Primeiro, uma enzima apelidada de “Dicer” corta qualquer RNA de cadeia dupla que encontra em pedaços que têm cerca de 22 nucleótidos de comprimento. Em seguida, complexos proteicos chamados RISC (complexo Silenciador induzido pelo RNA) se ligam aos fragmentos de RNA de cadeia dupla, o ventila, e então libera uma das cadeias, mantendo a outra., O complexo RISC-RNA então se liga a qualquer outro RNA viral com sequências nucleotídicas correspondentes às do RNA ligado ao complexo. Esta ligação bloqueia a tradução de proteínas virais pelo menos parcialmente, se não completamente. O sistema RNAi pode potencialmente ser usado para desenvolver tratamentos para genes defeituosos que causam a doença. O tratamento envolveria fazer um RNA de cadeia dupla a partir do gene doente e introduzi-lo nas células para silenciar a expressão desse gene., Para uma explicação ilustrada do RNAi, veja o módulo Flash curto e interativo em http://www.pbs.org/wgbh/nova/body/rnai-explained.html
o sistema de interferência de RNA também é explicado mais completamente no vídeo abaixo do Nature Video.
![]()
![]()
voltar ao topo | página anterior | próxima página